Osiągnięcie polskich naukowców przyspieszy prace nad nowymi terapiami z wykorzystaniem mRNA
Naukowcy z Uniwersytetu Warszawskiego opracowali nową metodę znakowania mRNA. Pozwoli ona na lepsze monitorowanie cząsteczek mRNA zarówno w komórkach, jak i w całym organizmie. Technologia, stworzona przez zespół pracujący pod kierunkiem prof. Jacka Jemielitego i dr hab. Joanny Kowalskiej, przyspieszy prace nad rozwojem terapii wykorzystujących mRNA.
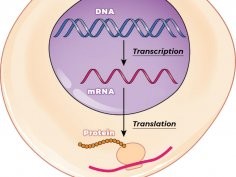
mRNA służą komórkom do produkcji białek.
Wytwarzanie białek przez komórki zaczyna się od skopiowania niewielkiego fragmentu DNA, czyli wykonania cząsteczki mRNA. Opuszcza ona jądro komórkowe i przechodzi do cytoplazmy. Tam przechwytują ją enzymy transportujące mRNA do rybosomów. W rybosomach mRNA jest odczytywane i na podstawie zawartych w nim instrukcji produkowane są cząstki białka. Cząstki mRNA są bardzo nietrwałe i szybko ulegają degradacji. Dlatego też naukowcy, pracujący nad wykorzystaniem mRNA w medycynie, musieli przezwyciężyć dwa problemy. Jednym z nich było ustabilizowanie mRNA tak, by zwiększyć jego odporność na obecne w komórkach czynniki degradujące. Z drugiej zaś – konieczne było zwiększenie jego produktywności, zatem by wykorzystywane w terapii mRNA częściej trafiało do rybosomów. Już w 2007 roku obie te trudności udało się przezwyciężyć zespołowi prof. Jemielitego i dr Kowalskiej.
Teraz, gdy technologie mRNA zaczynają być stosowane na szeroką skalę – tutaj przykładem są szczepionki przeciwko COVID-19 – i rośnie zainteresowanie tą technologią, pojawiło się kolejne wyzwanie. Naukowcy opracowujący nowe terapie z wykorzystaniem mRNA chcieliby mieć możliwość dokładnego śledzenia losów cząsteczek mRNA podanych do organizmu.
Naukowcy z UW dodali właśnie do jednego z końców mRNA, zwanego cap 3', znaczniki fluorescencyjne, pozwalające na monitorowanie cząsteczki w żywych komórkach. Co ważne, zastosowana przez Polaków technologia jest kompatybilna z tą stosowaną do oznaczania drugiego z końców, cap 5'. To zaś oznacza, że dzięki uczonym z Warszawy możliwe jest oznaczanie obu końców mRNA. Co więcej, każdy z nich może zostać oznaczony innym kolorem, a znaczniki mogą wymieniać między sobą energię jeśli znajdą się blisko siebie.
Podwójne znakowanie mRNA umożliwia zlokalizowanie tej cząsteczki w żywych organizmach. Nasze badania prowadzone były na organizmie kijanki Danio pręgowanego. W ich trakcie wykazaliśmy, że można prowadzić monitoring, czy i kiedy mRNA ulega degradacji, a także sprawdzać, jak efektywnie powstaje z niego białko. Podwójne wyznakowanie fluorescencyjne utrzymuje w pełni funkcjonalność mRNA, zarówno w liniach komórkowych, jak i żywym organizmie. W naszych badaniach wyznakowane mRNA odpowiadało za produkcję białka GFP. Jest to białko fluorescencyjne, które dawało światło w innym kolorze niż oba znaczniki zastosowane w mRNA. To pozwoliło nam jednocześnie obserwować losy wyznakowanego mRNA i powstającego białka. Co ważne, opracowana przez nas technologia pozwala tworzyć sondy molekularne, które umożliwiają monitorowanie enzymów odpowiedzialnych za metabolizm mRNA w komórkach żywych, stwierdził profesor Jemielity.



Komentarze (3)
lanceortega, 10 listopada 2021, 17:03
"Nasze badania prowadzone były na organizmie kijanki Danio pręgowanego."
Naprawdę? Danio pręgowany to rybka a nie płaz. Zatem ma stadium larwalne, ale nie jest to żadna kijanka.
Ergo Sum, 10 listopada 2021, 18:41
Są takie organizmy, które nadają się lepiej do badań z kilku względów - szybkości namnażania, małej ilości nieprzewidzianych mutacji oraz z powodu dużej bliskości do genomu człowieka - np. muszki owocowe są też takimi stworzeniami które mają bardzo dobre warunki. Innymi popularnymi są też oczywiście myszy - dlaczego nie np. świnki morskie? - z podobnych względów co danio pręgowane.
lanceortega, 11 listopada 2021, 11:48
Przecież chodziło mi o błędne użycie nazwy "kijanka"!
Kijanki są u płazów. Nie u ryb.