Sezonowe koronawirusy mutują w obrębie białka S. Będziemy musieli dostosowywać szczepionki?
Co najmniej od początku bieżącego roku media ekscytują się doniesieniami o pojawieniu się nowych wariantów wirusa SAR-CoV-2. Nie od dzisiaj wiemy, że wirusy mutują, więc nowych wariantów należało się spodziewać. Jednak zmiany mogą niepokoić, szczególnie jeśli wpływają na naszą pamięć immunologiczną lub skuteczność szczepionek. Naukowcy postanowili zbadać jakiego rodzaju zmiany zachodzą wśród koronawirusów atakujących ludzi od dziesiątków lat i dowiedzieć się, w jaki sposób mutacje SARS-CoV-2 mogą wpłynąć na przyszłe interakcje wirusa z człowiekiem.
Znamy setki koronawirusów krążących wśród nietoperzy, świń, wielbłądów czy kotów. Wiemy też, że 7 z nich atakuje ludzi. Cztery powodują objawy podobne do łagodnego przeziębienia i zarażają w sezonie grypowym kilkadziesiąt procent chorych. To sezonowe wirusy 229E, NL63, OC43 oraz HKU1. Trzy kolejne wirusy mogą powodować poważne zachorowania i prowadzić do zgonu: MERS-CoV, SARS-CoV oraz SARS-CoV-2.
Wiemy, że niektóre koronawirusy ponownie zarażają ludzi. Nie jest jednak jasne, czy jest to spowodowane dryfem genetycznym [czyli powolnymi mutacjami wirusa – red.] czy też utratą pamięci immunologicznej przez ludzki organizm. Chcieliśmy sprawdzić, czy istnieją jakieś dowody na to, że koronawirusy podobne do SARS-CoV-2 zmieniają się, by uniknąć ludzkiego układu odpornościowego, mówi doktor Kathryn Kistler z Wydziału Szczepionek i Chorób Zakaźnych we Fred Hutchinson Cancer Research Center w Seattle.
Naukowcy przyjrzeli się czterem koronawirusom wywołującym sezonowe przeziębienia. Wiemy, że wirusy te zidentyfikowano u ludzi 20–60 lat temu, wiemy, że ponownie infekują ludzi, nie wiemy jednak, czy przyczyną ponownych infekcji jest dryf genetyczny czy utrata przeciwciał.
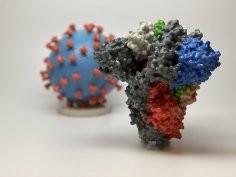
Uczeni użyli wielu różnych technik obliczeniowych, by przyjrzeć się ewolucji tych wirusów w czasie. Szczególnie interesowały ich zmiany, jakie mogły zajść w proteinach mogących zawierać antygeny, czyli np. w białku S, które znajduje się na powierzchni wirusa i jest tym samym wystawione na działanie układu odpornościowego.
Okazało się, że u dwóch koronawirusów – OC43 i 229E – mamy do czynienia z szybkim tempem ewolucji białka S. Niemal wszystkie mutacje, które są korzystne dla tych wirusów, zaszły w regionie S1 tego białka. To właśnie ten region pomaga w infekowaniu komórek ludzkiego organizmu.
Wyniki badań wskazują, że wirusy te ulegają szybkiemu dryfowi genetycznemu, by uniknąć układu odpornościowego. Co więcej, z przeprowadzonych szacunków wynika, że przydatne wirusowi mutacje białka S (białka kolca) OC43 i 229E pojawiają się raz na 2-3 lata. To mniej więcej dwu- a nawet trzykrotnie szybciej niż mutacje obserwowane w wirusie grypy H3N2.
W związku z dużą złożonością i zróżnicowaniem sezonowych koronawirusów, nie jest do końca jasne, czy koronawirusy takie jak SARS-CoV-2 również ewoluują w ten sam sposób. Może się okazać, że co jakiś czas trzeba będzie zmieniać obecnie stosowane szczepionki przeciwko COVID-19 tak, by zwalczały nowe szczepy SARS-CoV-2. Kluczowym elementem walki z tą chorobą będzie więc ciągłe monitorowanie ewolucji antygenów wirusa, dodaje Trevor Bedford, główny autor badań.
Ze szczegółami pracy uczonych z Seattle można zapoznać się na łamach eLife.



Komentarze (28)
mezizen, 21 stycznia 2021, 11:18
Jeszcze nikt wirusa nie wyizolował, a już wiedzą, że mutuje. Przy takiej "medycynie" znachorstwo jest bardziej godne zaufania.
Mariusz Błoński, 21 stycznia 2021, 11:33
Poniżej linki z namiarami na frajerów, którym wydawało się, że wyizolowali. Proponuję napisać maila i wyprowadzić ich z błędu:
https://www.ncbi.nlm.nih.gov/pmc/articles/PMC7239045/
https://jvi.asm.org/content/94/11/e00543-20
https://www.ncbi.nlm.nih.gov/pmc/articles/PMC7036342/
cyjanobakteria, 21 stycznia 2021, 15:28
Czasami zdarza się, że medycyna jest faktycznie bezradna. Może przyłóż sobie świeże liście babki lancetowatej do czoła, a najlepiej całą główkę kapusty. Inteligencja się co prawda nie sumuje, ale na pewno nie zaszkodzi. Kto wie, może dojdzie do tak zwanej zimnej fuzji?
mezizen, 21 stycznia 2021, 15:33
Jeżeli mogą wyizolować, to dlaczego testują metodą PCR, która nie nadaje się do diagnostyki?
Czy Ty cjanobakteria tylko tak niekostruktuwnie lubisz sobie pisać czy rzeczywiście znasz się na znachorstwie i na co pomogła Ci ta babka?
cyjanobakteria, 21 stycznia 2021, 15:56
Jestem ekspertem w znachorstwie. Mi na rozum nie pomogło, ale mokre liście trochę ekranują od 5G
Za to twoje dotychczasowe kontrybucje są legendarne.
mezizen, 21 stycznia 2021, 16:00
Uważam cynizm za bezsensowny, ale mimo wszystko życzę miłego dnia.
cyjanobakteria, 21 stycznia 2021, 18:57
To był sarkazm, ale sądząc po twojej dotychczasowej aktywności na forum, raczej się nie dogadamy.
mezizen, 21 stycznia 2021, 19:26
Cjanobakteria, jeżeli sarkazm nie jest Twoją typową reakcją na poglądy, z którymi się nie zgadzasz, to pochopnie oceniłem Cię jako cynika. Wybacz.
Czy @Antylogik został zablokowany na forum, że się go o to podejrzewa?
Przypuszczam zatem, że ma poglądy podobne do moich w temacie rzekomego wyizolowania wirusa i testów PCR, które nie są przeznaczone do diagnostyki? Jakie jest Twoje @Astro zdanie w tej kwestii?
cyjanobakteria, 21 stycznia 2021, 19:50
Reaguje tak tylko na wpisy wietrzących teorię spiskową. Jeżeli jesteś tylko niedoinformowany albo masz zły dzień, to pochopnie oceniłem. Wybacz.
mezizen, 21 stycznia 2021, 19:58
Rozumiem. Nie każdy jest w stanie zmieniać swoje poglądy, a nawet może nie chcieć o nich dyskutować, jeżeli uważa swoje za ugruntowane a innych za należące do niepopularnych czy jak je nazywasz "teorii spiskowych".
Pozwolę sobie zapytać, czy Ty masz lub miałeś w swoim życiu jakiś niepopularny pogląd?
mezizen, 21 stycznia 2021, 20:17
Najpierw jest pogląd / idea, za nim idzie doświadczenie, potem doświadczenie powtarzalne, potem dopiero fakty. Jeżeli fakty są wynikiem błędu czy fałszu doświadczenia, to już nie są faktami, tylko błędem lub oszustwem.
Jak przeprowadzono niepodważalne doświadczenie na wyizolowanie wirusa powodującego COVID-19, jeżeli nie ma wiarygodnego testu diagnozującego COVID-19? Mam nadzieję, że zgadzasz się chociaż z tym, że test PCR nie nadaje się do diagnostyki?
mezizen, 21 stycznia 2021, 20:25
Jeżeli się na czymś nie znasz, warto zasięgnąć do źródła. Łatwo znajdziesz publikacje, a nawet wywiady z wynalazcą technologii PCR, które mówią o nie nadawaniu się tej technologii do diagnostyki. Czytałeś chociażby instrukcję testu PCR? Tam też o tym piszą...
mezizen, 21 stycznia 2021, 20:28
Ty nie "plusujesz" wiadomości, z którymi się zgadzasz?
Uściślij proszę lub zadaj pytanie ponownie, żeby wiedział o co dokładnie pytasz. Postaram się odpowiedzieć.
Moja wątpliwość odnośnie wiarygodności testów PCR też jest wciąż otwarta.
mezizen, 21 stycznia 2021, 20:46
Astro, masz ideę, doświadczenia potwierdzającego Twój pogląd jeszcze nie masz. Co tu dopiero mówić o fakcie. Mam nadzieję, że nie jesteś naukowcem. (nie atakuję Cię, po prostu szukam logiki w Twojej komunikacji)
Jeżeli uważam pracę tych ludzi za nielogiczne, to jaki sens ma ich uwzględnianie. Autorytetu u mnie nie mają gdyż opierają swoją prace na błędnych przesłankach - niepodważalności testu PCR.
Czy ktoś mi w końcu odpowie jak można wyizolować przyczynę efektu E, jeżeli nie ma niepodważalnej metody sprawdzenia czy to jest właśnie to E?
Jajcenty, 21 stycznia 2021, 21:16
Hm, z czego zatem zrobiona jest Sputnik V i ta tajemnicza chińska? Rozumiem, że Pfizer i reszta zgadli sekwencję aminokwasów. Przypominam że skuteczność wszystkich tych preparatów jest większa niż 90% więc trudno to jakoś uznać za przypadek. IMHO, musieli mieć przynajmniej jednego wirusa do dyspozycji.
lanceortega, 22 stycznia 2021, 13:02
Dokładna sekwencja genetyczna (przepisana na DNA w procesie RT-PCR) wszystkich znanych wariantów SARS-CoV-2 jest znana i udostępniona w publicznej domenie. Wystarczy kilka minut, aby sekwencję znaleźć. Po co te brednie że nie wyizolowano wirusa?!
Przykład:
https://www.ncbi.nlm.nih.gov/nuccore/MN988668
Jajcenty, 22 stycznia 2021, 14:49
Strasznie się jąka...
cyjanobakteria, 22 stycznia 2021, 14:54
Dysponujemy portretem pamięciowym
https://www.news-medical.net/news/20200610/Asymptomatic-spread-of-SARS-CoV-2-appears-to-be-rare-says-WHO.aspx
Pii, 26 stycznia 2021, 21:33
Oj cyjanobakterio. Dotychczas te niedobre wirusy były utrzymane w kolorystyce czerwono zielonej (zobacz na swoje źródło) A tu nagle takie piękne kolorki fotoszopowy wirusolog namalował. Czy to już jest ten postęp co on wziął się i nadszedł po zaprzysiężeniu kierownika budowy demokracji takiej bez widowni pośród 200 tyś flag? Czy dopiero przełom nastąpi po tym gdy wirusy staną w pełni tęczowe?
cyjanobakteria, 26 stycznia 2021, 22:13
Pewnie ta wiedza ci się do niczego nie przyda, ale obrazy z mikroskopu elektronowego, SPM i bardziej zaawansowanymi instrumentów nie zawierają kolorów, więc wszystkie są kolorowane ręcznie, być może nawet w fotoszopie. Sugeruję zmianę lekarza prowadzącego
Tu masz wirusa w kolorach smerfetki w czerwonych szpilkach.
https://www.biorxiv.org/content/10.1101/2020.08.20.259531v1.full
l_smolinski, 29 stycznia 2021, 09:09
Nie ma szans na na logikę. Z tym panem to się po prostu nie uda.
peceed, 14 kwietnia 2021, 08:14
To ewolucja a nie dryf genetyczny. Dryf genetyczny z założenia nie wpływa w znany sposób na fitness.
Jarek Duda, 10 lipca 2021, 12:30
Ciekawe porównanie: https://www.businessinsider.com/cdc-covid-19-surges-counties-low-vaccination-rates-93-40-2021-7?IR=T
A tymczasem w Polsce: https://dziennikpolski24.pl/szczepienia-przeciwko-koronawirusowi-w-polsce-10072021-jak-wyglada-sytuacja-w-kraju/ar/c14p1-21791559
peceed, 10 lipca 2021, 16:54
Polityczna manipulacja a do tego fałszywka która ma na celu dokopanie Teksasowi/Republikanom?
Przede wszystkim zaznaczone hrabstwa na prawej mapie muszą być podzbiorem tych na lewej, a jest odwrotnie, z jednostkowymi wyjątkami (fałszerstwo).
A samo sformułowanie może sugerować znacznie większą korelację tych dwóch wielkości niż istnieje w rzeczywistości (manipulacja).